Summary of IVF mosaic embryo project.
Our research work evaluating the safety of mosaic IVF embryo transfer has been published recently. Here I briefly summarized research progress of this project.
IVF嵌合胚胎课题是我来汤组之后最早接手的两个课题之一,也是第一篇由我独立负责生信分析的研究论文,课题体量和难度都不大,算是典型的短平快课题了。但得益于实验样本的珍贵性、科学问题的重要性、以及单细胞多组学技术的优越性,这个课题起初被大家基于厚望。然而课题过程却并不顺利:疫情带来的取样困难问题、非侵入取样带来的数据质量问题、最后投稿时被抢发论文冲击的问题,都给课题的顺利推进与发表带来了不小的困难。最终课题发表在Genomics, Proteomics & Bioinformatics上,也算一个圆满的结局吧。
课题的研究思路、实验设计、结果结论和科学意义都可以参考原文链接以及新闻稿 (English version),就不再自己总结了,在这里主要想记录一下在课题推进中的各种经历与心路历程。
课题过程
课题开始于2020年9月。9月8日高原师姐在汤老师的推荐下联系了我,提到与301医院姚老师合作了一个课题,主要内容是新生儿DNA与RNA的双组学测序分析,问我是否愿意合作。我欣然答应,并着手复现舒惠师姐留下的数据与分析结果。值得一提的是,我来到汤组报道是当年的9月10日,在接手课题时其实与实验室的大家并不相识,这里要感谢汤老师和师姐对我的信任,把课题的分析任务交给了我这个还没进入大四的本科生。
到实验室报道后,申请超算账号,搭建工作环境,找师姐了解课题背景,为之后的工作做准备。到了18号,第一批测试数据释放,对建库流程和计算方法进行了验证。11月2号上机三个正常小朋友的样本,拿到结果和文老师讨论。之后便进入了漫长的等待样品时期,4月1号拿到第一个异常胚胎的样本,再到7月13日收齐所有8个异常胚胎样本,中间经历了疫情反复、取样不便等诸多困难,收样过程就经历了大半年。
收齐所有数据后便开始集中分析,测试了一批scCNV的方法,最终拿到了认为稳定的结果,并在汤老师的建议下进行了多轮横向与纵向的交叉验证,最终形成结果结论,由师姐动手主导完成论文写作。12月7日第一次投稿NEJM、被拒,1月13日第二次投稿 Nature Medicine、送审被拒,3月8日第三次投稿Fertility and Sterility、送审被拒,再到4月19日投稿GPB,7月26日接收,投稿过程历时7个多月,也算是历经坎坷。
另外插一句,刚刚来的时候师姐跟我聊天好像是带小朋友哈哈哈哈哈,当时真的初来乍到,对实验室、课题本身、计算流程没有那么熟悉,也没有专门的师兄师姐来带我,就一点一点摸索一点一点成长,是很有意思的经历!
一些困难
TSO串联与ploy-G
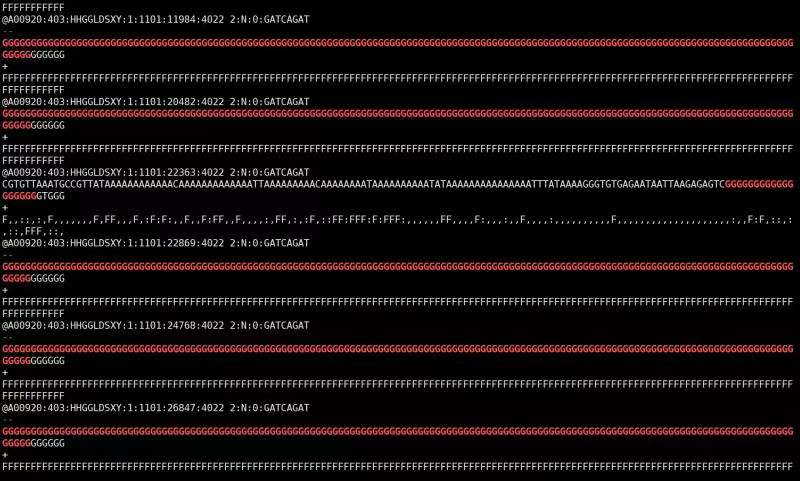
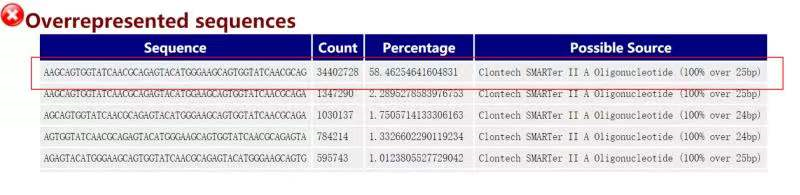
印象很深的是当时第一批测试小鼠数据质量并不太好,仔细check一下之后发现数据R1出现了比较强的TSO串联(>50%),以及R2上不明来源的poly-G序列,至今没有找到非常合理的解释,只能归因于细胞质量不好。当时请教了森和其他师兄师姐,大家均表示没有遇到类似的情况,但我们的结果中这些异常是文件的。有趣的是,之后大家做STRT-seq的单细胞RNA都先后出现了类似的情况,而且都是在我提出之后才发现了类似的问题。没想到这么一个全组普遍存在的问题竟然被我一个最低年级的同学率先发现了,还是有些神奇。
问题的解决也十分神奇,或者说问题根本没有得到解决,只是我们的课题设计仅仅需要scRNA进行细胞类型鉴定,并不需要很高质量与灵敏度的单细胞数据。而之后的数据中串联情况有所好转,便保留下来了。
scCNV计算流程
之后便是测试数据的单细胞CNV测试。由于建库流程直接使用了周圆师姐之前的protocol,计算上便也直接使用了舒惠师姐留下的scMALBAC以及scCNV流程,用gingko流程推测单细胞CNV。然而gingko主打的还是web server,真正的命令行工具并不好用。下载下来的代码明显只支持SGE文件系统的服务器,代码里面qsub嵌套着qsub,在北极星的slurm上一跑直接给投到GPU节点上去了,吓得赶紧scancel。之后又经历了漫长的流程修改,把qsub一个一个按逻辑改成sbatch,然后一点点修改各种目录结构,光改代码就好几天才跑通。
之后的另一个问题是ginkgo只提供了计算好的hg19基因组,没有hg38。原本的思路是利用它内嵌的buildGenome脚本自己写一个hg38,但这个结构更复杂……只记得大四的我调了一整天也没调通,最后放弃,老老实实用旧版基因组hg19。不过后来发现他们TE活检的结果也是hg19的,到也算是不谋而合了哈哈哈哈,省下了之后liftover的功夫。
值得一提的是,后来弃船FreeC投奔ginkgo的慧经过复杂的调试,成功构建了gingko所需的hg38基因组的文件,属实是居功至伟。之后再做单细胞CNV也许可以找慧慧索取一下哈哈哈。
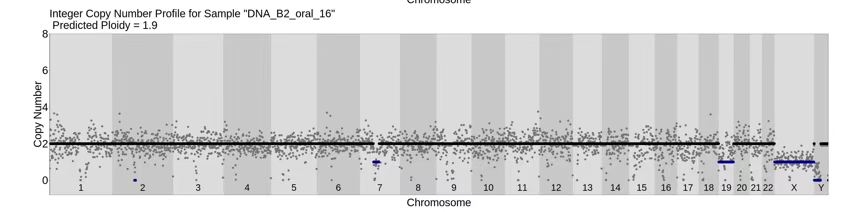
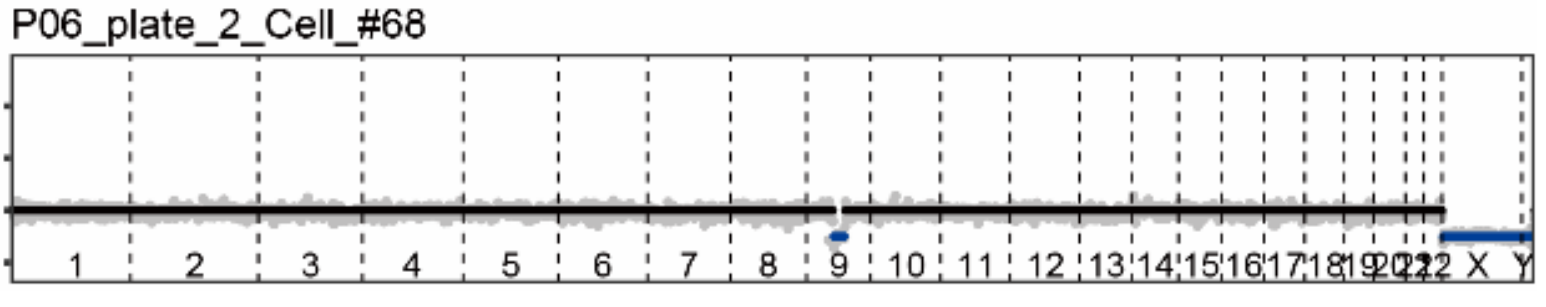
搞定命令行软件之后进行参数调试,刚刚开始的方法效果似乎并不美好,算出来正常细胞的CV高出天际。之后也试了很多办法,首先是使用正常细胞pool在一起当做control,以消除MALBAC扩增的偏好性。结果似乎稍微好了一点,但仍然不够让人满意。挣扎一番之后甚至动了弃船的念头,想去投靠control-FreeC流派。最后耐着性子去读了gingko的源码,突然发现它散点图(cloud)用的数据是normalize control之前的……我当场昏厥。于是又在可视化代码里面做了一点改动,把散点图的纵坐标换成了normalize之后的数据,果然效果好了不少。不禁感叹这种坑真的就只有读了源码才知道啊,加上作者的变量命名一塌糊涂……用了很多意义不明的变量名,读来真的折磨。
另外一个有意思的发现是MALBAC的扩增偏好性也存在不小的批次效应,具体表现一批样本的数据产生的merge control并不适用于另一个样本。遂就此修改了设计merge control的策略,将每个index下的96个细胞pooling在一起作为这个index的control,最终拿到了稳定且优质的结果,算是给scCNV画上一个句号。
备份与备案
备份与备案算是科研之外的程序了,备份程序还比较简单,上传数据或者硬盘拷贝,之后再网页填写就好了,时间周期也比较短。但备案就是另外一个故事了,只记得当时跑了很多很多次医学部,甚至最终掌握了从三院小门混进医学部的非法技能哈哈哈哈。总之过程就是非常繁琐,很多流程必须线下完成,这在疫情封校的当下似乎并不是一个容易的事情。
竞争与抢发
在我们进行第一版投稿的时候,另一篇研究相似问题的论文在The American Journal of Human Genetics杂志见刊了。他们使用SNP array进行了CNV的鉴定,得到的结论与我们相同。而他们的投稿时间是2021年9月15日,比我们提早了两个多月。总体来说他们的规模比我们更大,但是技术精度不及我们。然而他们抢先发表了,那我们研究的新颖性就大打折扣了。事实且确是如此,多个独立审稿人都提到了21年12月发表的那篇文章,认为我们的结果是对他们结论的“高精度独立验证”,只是提供了新的高可信度的证据,但结论本身不具有新颖性。也正因如此在投稿过程中遇到了不小的困难,最终发表在GPB上。但其实回头想想,我们课题开展更早,技术优势更大,且在课题推进中无论是师姐、合作方还是我都没有耽搁。然而人算不及天算,新冠疫情给取样带来了较大的困难,8个病人的样本经历了八九个月的安排协商才最终收齐,最终成果发表慢人一步。不过也好,我们既严格尊重了国家的防疫政策,没有给国家添麻烦,又最终响应了总书记“把论文发表在祖国大地上”的号召,也算是功德圆满。
临近中秋,新生返校加上海淀聚集性疫情,刚打开没多久的校门又被关上了。我已经不记得2022年的9个月份中能出校的日子有多少个,也不记得这半年不足两百日的光景中测过多少次核酸。我们一次一次被迫通过检测证明自己没病,又一次一次尝试使用“非必要”的理由找回自己的生活,最终都只不过是宏大叙事下微不足道的尘埃。然而这无足轻重的一丝尘埃,就已经是我们当下生活的全部。
睡吧,睡过这夏末秋初闷热聒噪的夜,睡到深秋寒凉、收成落定之时,应是另一番模样。
振宇
于畅春新园
2022年9月8日夜